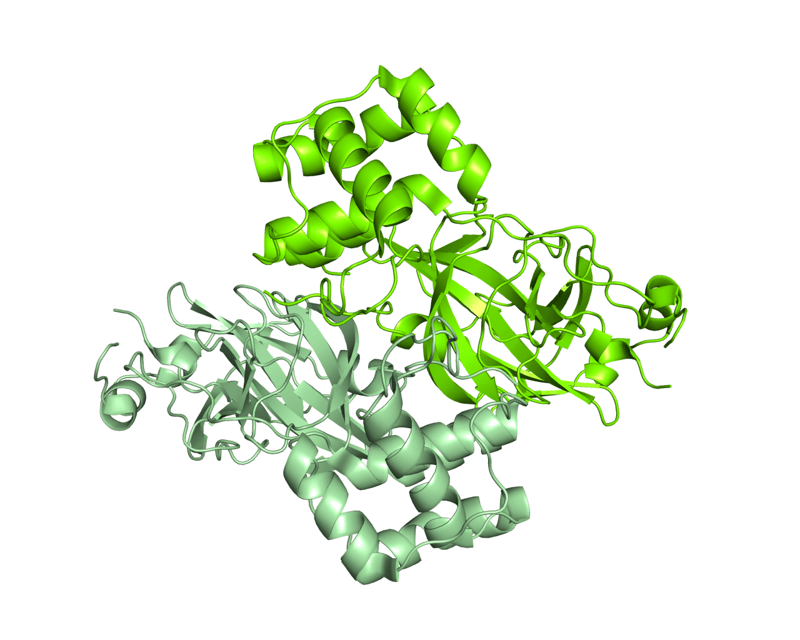Quante volte abbiamo sentito in questi mesi auspicare che vengano sviluppati antivirali specifici contro il SARS-CoV-2? Ovviamente – e giustamente – tante. Ma come si può fare perché questo auspicio diventi realtà? Quali strategie possiamo usare, sfruttando le informazioni che derivano dall’isolamento e sequenziamento del materiale genetico del virus?
Identificare il bersaglio molecolare
Per prima cosa occorre identificare un bersaglio molecolare verso cui puntare le nostre armi. E qui ci viene in aiuto la conoscenza pregressa della biologia dei Coronavirus.
Piccolo inciso, fino a venti anni fa i Coronavirus erano considerati poco pericolosi e chi li studiava sembrava interessarsi a un problema con scarse applicazioni per la salute umana. È proprio grazie allo studio guidato dalla curiosità di tanti scienziati, la ricerca di base, che oggi abbiamo una bussola per saperci come muovere nella battaglia contro il virus.
Quando i Coronavirus infettano le cellule, essi rilasciano il loro materiale genetico (una grande molecola di RNA) e questo viene utilizzato dai nostri macchinari cellulari – letteralmente dirottati – per sintetizzare le proteine del virus.
Per come è organizzato il virus stesso, alcune delle sue proteine vengono sintetizzate unite tra loro come perle in una collana. Ma per formare nuove particelle virali è necessario che le proteine si stacchino le une dalle altre. E qui intervengono delle forbici molecolari, le proteasi del virus, che tagliando la collana in punti ben specifici, liberano le proteine per consentire a ciascuna di svolgere la sua funzione.
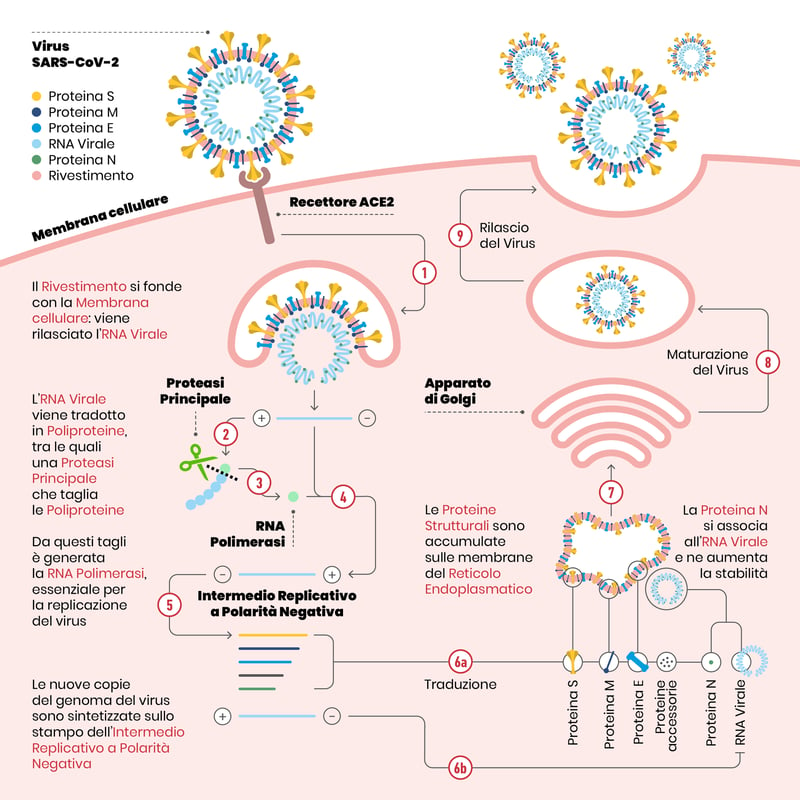
Schema che illustra il ciclo replicativo di SARS-CoV-2
La proteasi principale del SARS-CoV-2 si chiama Mpro (dall’inglese “Main Protease”), e senza la sua azione il virus non può replicarsi e infettare nuove cellule! Quindi una molecola che in pratica “spunti” queste forbici del virus sarebbe un composto con potenziale azione antivirale.
Questi composti si chiamano inibitori in quanto bloccano l’attività naturale della proteasi.
A rendere la Mpro un bersaglio ancora più attraente c’è la sua unicità, ovverosia il fatto che nell’organismo umano non ci sono proteine con una struttura e funzione analoga, per cui un composto che inibisca l’azione della proteasi virale avrebbe minori probabilità di avere effetti collaterali gravi sul paziente.
Ingegnerizzare le molecole
Ma come facciamo a ingegnerizzare una molecola che inibisca Mpro? Se ci state seguendo in questa serie, oramai la risposta la sapete: niente di meglio che visualizzare la struttura della proteina, vedere quale porzione di essa costituisce la “lama” delle forbici e che forma abbia per costruire qualcosa che impedisca che essa possa svolgere i suoi tagli molecolari.
Quello che vogliamo è ottenere un composto che abbia alta affinità verso la proteina, cioè che sia in grado di inibire Mpro, anche se presente in piccole quantità. Inoltre, è importante che abbia elevata specificità, evitando cioè che non venga distratto nel suo compito da altre proteine nelle cellule.
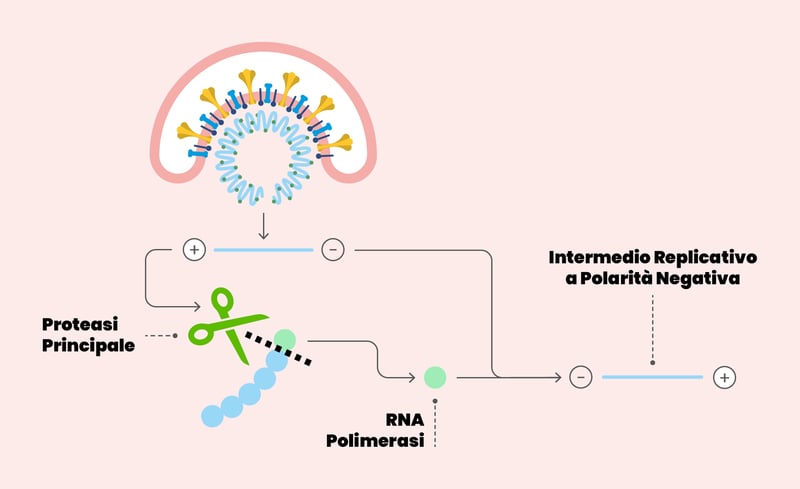
Dettaglio del ciclo replicativo di SARS-CoV-2. Il rivestimento del virus si fonde con la membrana cellulare. Una volta che l’RNA è stato rilasciato nel citoplasma, viene tradotto in poliproteine, tra le quali la proteasi principale (indicata nello schema con una forbice). La proteasi opera dei tagli sulle poliproteine: da uno si questi tagli si origina l’enzima RNA polimerasi, essenziale per la replicazione del virus.
Già nel mese di febbraio i gruppi di ricerca dei prof. Rao e Hilgenfeld hanno determinato le prime strutture di Mpro da SARS-CoV-2 [1] sia nella sua forma di “forbice aperta” che legata a diversi inibitori, uno dei quali altamente specifico e con elevate potenzialità di rappresentare un futuro antivirale [2].
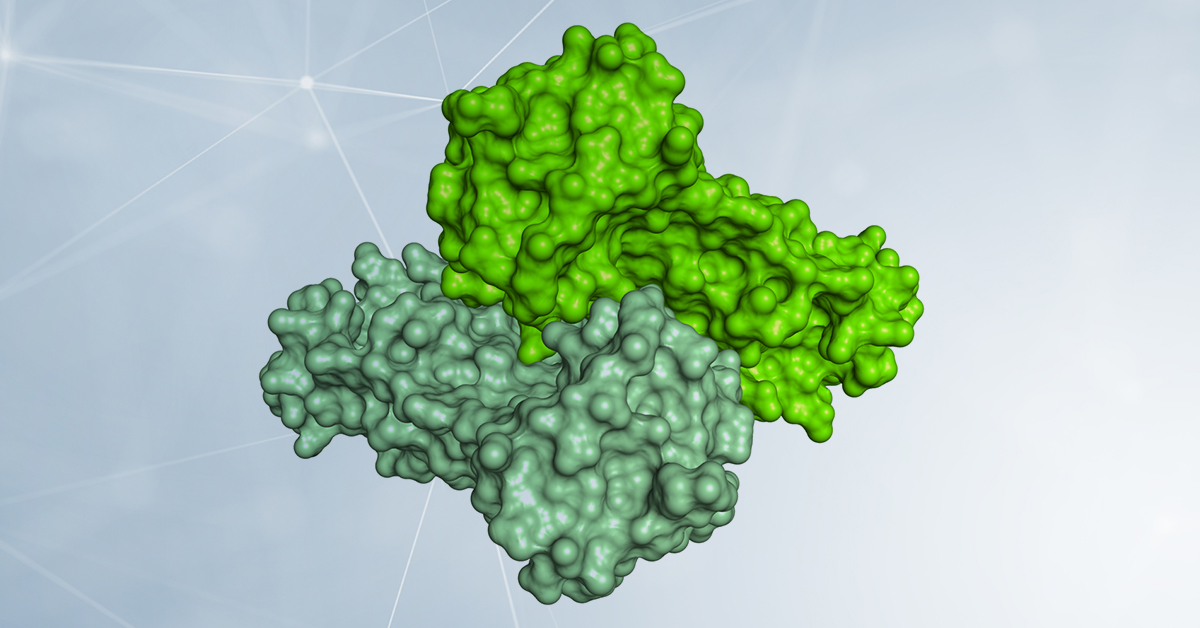
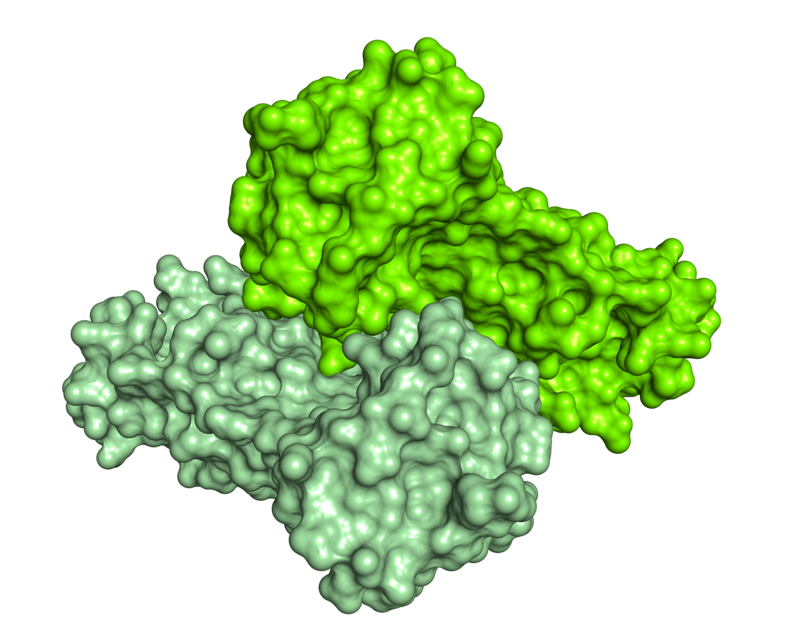
La proteasi Mpro è un dimero, ossia per funzionare dev’essere formata da due catene uguali associate tra loro, e ha una tasca superficiale in cui avviene il taglio della poliproteina virale. Le caratteristiche di questa tasca (forma, dimensioni) vanno sfruttate per ingegnerizzare un inibitore ottimale.
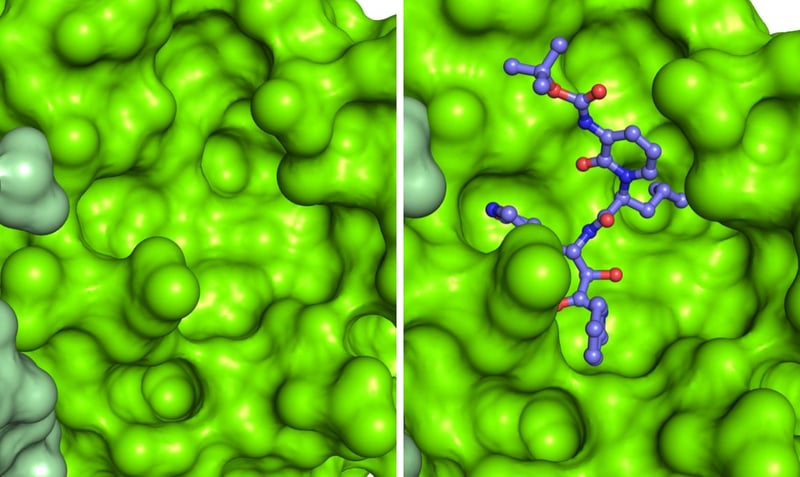
Dettaglio della struttura dimerica di Mpro da SARS-CoV-2, rappresentata come superficie molecolare, con le due catene colorate in due tonalità di verde. L’analisi delle caratteristiche strutturali della proteina ha consentito di identificare una tasca superficiale in cui avviene il taglio della poliproteina virale, e di ingegnerizzare inibitori. Un esempio è rappresentato da un composto che è complementare come forma e caratteristiche chimiche alla tasca superficiale e che inibisce l’attività di Mpro. La struttura utilizzata per realizzare le immagini è depositata con il codice 6Y2F nel Protein Data Bank (http://www.rcsb.org/pdb). Per gentile concessione del Dott. Degano.
In totale, al momento, sono state determinate 152 strutture di Mpro legata a diversi frammenti di molecole che danno indicazioni su come migliorare l’affinità e la specificità degli inibitori. In questo caso la tecnica utilizzata è quella della cristallografia a raggi X, una tecnica che dal 1945 continua a consentirci di visualizzare molecole biologiche con un dettaglio fino a livello del singolo atomo. Nella prossima puntata vedremo cos’è e come funziona la cristallografia!
Citazioni
[1] Jin et al. “Structure of Mpro from SARS-CoV-2 and discovery of its inhibitors” (2020) Nature 582, 289-293. DOI: https://doi.org/10.1038/s41586-020-2223-y
[2] Zhang et al. “Crystal structure of SARS-CoV-2 main protease provides a basis for design of improved α-ketoamide inhibitors” (2020) Science 368, 409-412. DOI: 10.1126/science.abb3405