La pandemia in corso di COVID-19 ha imposto alla comunità scientifica di dedicarsi alla ricerca e sviluppo di varie strategie per combattere l’infezione da SARS-CoV-2. L’unico modo informato per identificare nuovi antivirali passa attraverso la conoscenza della struttura tridimensionale, letteralmente la forma, delle proteine del virus che sono responsabili dell’infezione delle cellule e della replicazione del virus.
Una volta nota la struttura delle proteine, è possibile per i chimici sintetizzare dei composti che ne bloccano la funzione, e di conseguenza il ciclo vitale del virus.
Ne parliamo in questo ciclo di 5 puntate con l’aiuto del Dott. Massimo Degano, Group leader dell’Unità di Biocristallografia dell’IRCCS Ospedale San Raffaele e docente presso il nostro Ateneo (insegna Chimica al Corso di Laurea in Odontoiatria e Protesi dentaria e all’International MD Program, Biochimica al Corso di Laurea in Medicina, e Biologia strutturale al Corso di Laurea in Ricerca Biotecnologica in Medicina).
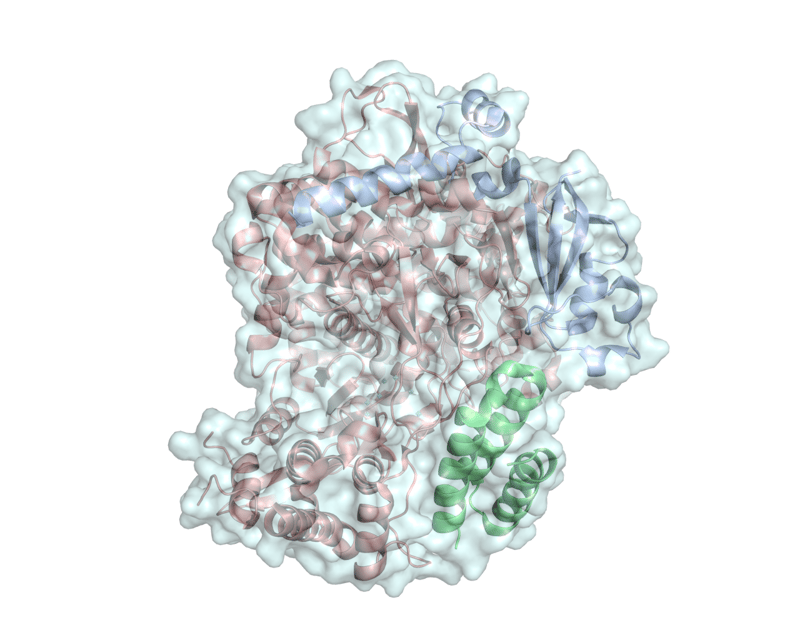 La struttura della RNA polimerasi di SARS-CoV-2, composta di tre subunità proteiche a formare un eterotrimero attivo. Gentile concessione del Dott. Degano.
La struttura della RNA polimerasi di SARS-CoV-2, composta di tre subunità proteiche a formare un eterotrimero attivo. Gentile concessione del Dott. Degano.
L’infezione da SARS-CoV-2 e la sua RNA polimerasi
Come abbiamo visto precedentemente (leggi l'articolo Sviluppare antivirali in modo razionale: “spuntare” le forbici di SARS-CoV-2), quando avviene un’infezione virale, il virus entra all’interno della cellula ospite, inizialmente agganciandosi tramite un recettore virale (nel caso di SARS-CoV-2, la proteina Spike) che si attacca a una proteina della cellula bersaglio (per SARS-CoV-2, l’enzima ACE2).
Una volta all’interno della parte acquosa della cellula, chiamata citosol, il virus si “scompone” rilasciando il suo contenuto: un misto di proteine e del suo materiale genetico.
Mentre nell’uomo l’informazione genetica, cioè quella che serve a costruire le cellule, è conservata in molecole di DNA, il Coronavirus utilizza una singola molecola di RNA. Anche gli umani hanno RNA nelle loro cellule, ma a noi serve per altri scopi, in primis per costruire le proteine.
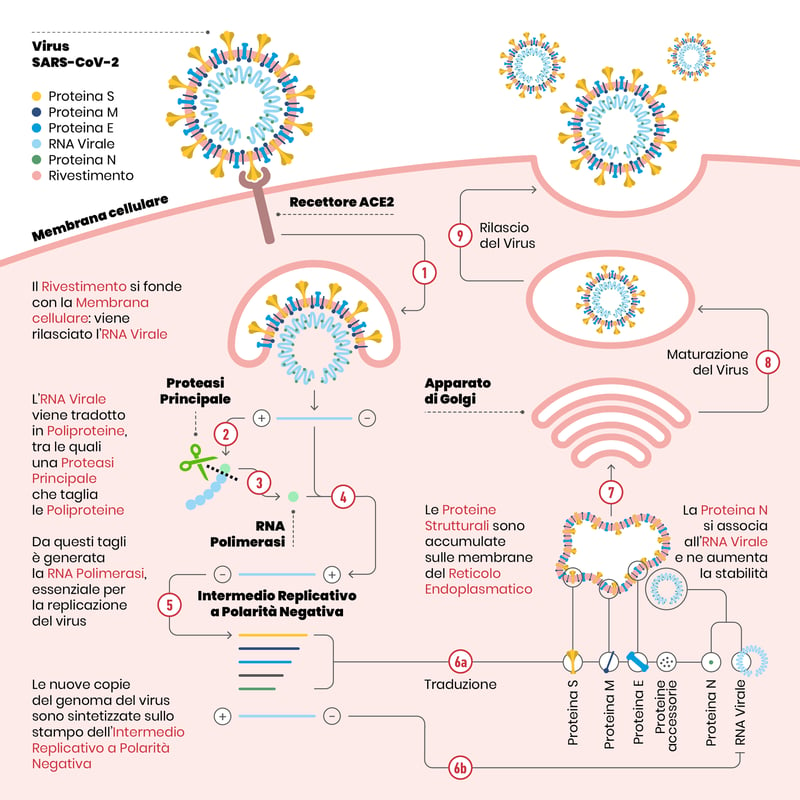
Schema che illustra il ciclo replicativo di SARS-CoV-2
Una volta che il coronavirus ha infettato una cellula, il suo scopo è far sì che si costituiscano nuove copie del virus stesso, in modo da avere una progenie in grado di perpetuare la specie.
Per cui, tutto quello che era presente nel virus che ha infettato la cellula dev’essere “copiato” e assemblato per formare nuovi virioni. E per questo ci vogliono nuove copie del materiale genetico (=RNA) del virus stesso.
Il macchinario essenziale per sintetizzare nuovo RNA si trova, in diverse forme, in tutti gli organismi e si chiama RNA polimerasi, letteralmente: “enzima che forma lunghe catene di RNA”.
La RNA polimerasi di SARS-CoV-2 si chiama RdRP (RNA polimerasi RNA-dipendente, dall’inglese RNA-dependent RNA polymerase) e in pratica copia il materiale genetico del virus.
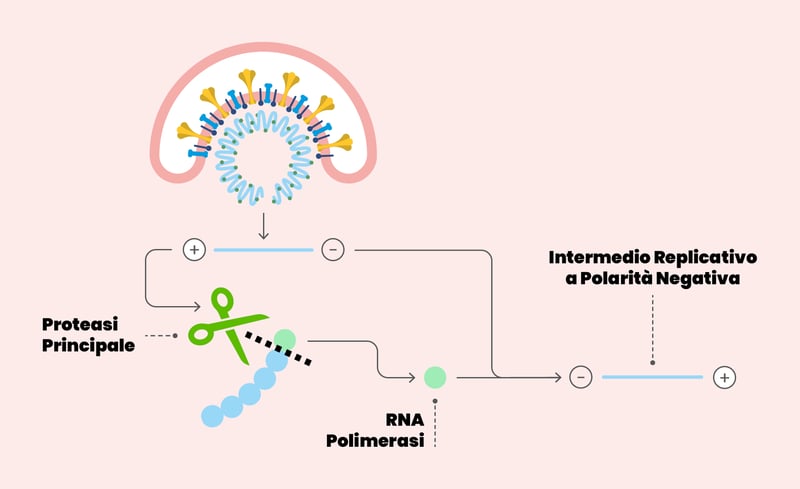
Dettaglio del ciclo replicativo di SARS-CoV-2. Il rivestimento del virus si fonde con la membrana cellulare. Una volta che l’RNA è stato rilasciato nel citoplasma, viene tradotto in poliproteine, tra le quali la proteasi principale (indicata nello schema con una forbice). La proteasi opera dei tagli sulle poliproteine: da uno si questi tagli si origina l’enzima RNA polimerasi, essenziale per la replicazione del virus.
Una “fotocopiatrice distratta”
Ma il macchinario (o l’operatore) è “distratto”, infatti ogni volta che fa una copia del materiale genetico originale, commette degli errori, e la copia presenta delle differenze rispetto all’originale.
Queste differenze sono le cosiddette mutazioni che si ripercuotono sulla struttura delle proteine del virus e sulla loro funzione. In questi giorni abbiamo sentito parlare della variante G614 (o meglio, D614G) del virus: bene, quello che è successo è che l’RNA polimerasi si è “distratta” facendo una copia del genoma virale, e ha cambiato una base (un “mattoncino”) di RNA.
Questo ha causato una mutazione della proteina Spike nella posizione 614, che però non è un sito di contatto diretto con l’enzima ACE2, per cui l’effetto sulla struttura e funzione dovrà essere valutato sperimentalmente per capirne le conseguenze.
In ogni caso, queste mutazioni hanno delle conseguenze sul virus, cioè possono essere neutre, vantaggiose o svantaggiose.
Un virus mutato inizierà a circolare se questa mutazione gli dà un vantaggio per il suo ciclo vitale: questo può voler dire essere più abile nell’infettare cellule umane, ad esempio, ma provocare meno danni nell’organismo ospite, dato che un virus altamente letale è destinato a scomparire per mancanza di ospiti.
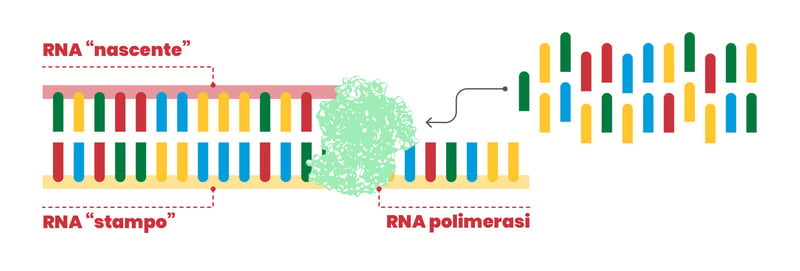
L’enzima RdRP sintetizza una copia del materiale genetico del virus, una molecola di RNA composta da quattro diverse basi azotate. L’enzima prende il filamento di RNA da copiare e appaia ad esso, una alla volta, le singole unità molecolari (i nucleotidi) ad esso complementari secondo l’appaiamento A:U e G:C (in figura, i rettangoli con colori diversi si appaiano giallo con blu e rosso con verde). A volte RdRP sbaglia appaiamento, e questo porta a una mutazione nella “progenie” virale. Se la mutazione è vantaggiosa per la sopravvivenza, essa verrà mantenuta.
L’antivirale Remdesivir
Torniamo alla funzione primaria della RNA polimerasi, ossia copiare il materiale genetico di SARS-CoV-2.
Questo è un passaggio basilare per formare nuovo virus, e quindi se riuscissimo a bloccare l’azione di RdRP nelle cellule infettate… risposta esatta, il virus smetterebbe di replicarsi. Molecole in grado di inibire la RNA polimerasi sono ottimi antivirali!
E la molecola Remdesivir, sviluppata per inibire l’enzima RNA polimerasi del virus dell’Ebola ma in grado di bloccare anche le polimerasi di altri virus, è un ottimo esempio di antivirale con bersaglio RdRP.
Analogamente a quanto visto per la proteasi principale Mpro di SARS-CoV-2, la biologia strutturale può dirci come fa Remdesivir a inceppare il macchinario, e soprattutto suggerirci come migliorarla per far sì che si possa ottenere un antivirale altamente specifico per il coronavirus.
Ricercatori dell’Accademia delle Scienze Cinese hanno determinato, usando la tecnica della criomicroscopia elettronica, la struttura di RdRP, fotografandola nel bel mezzo della sua attività replicativa [1].
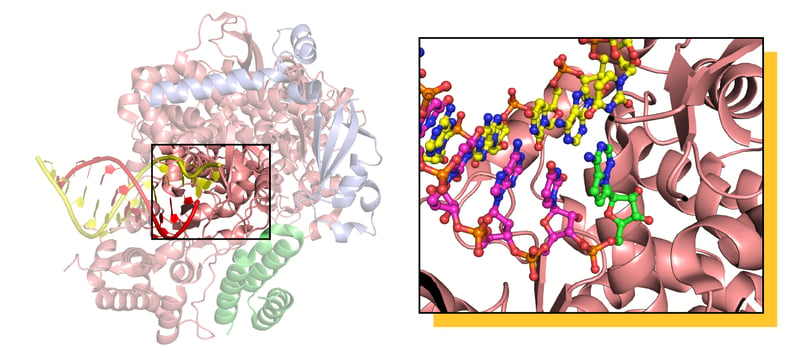
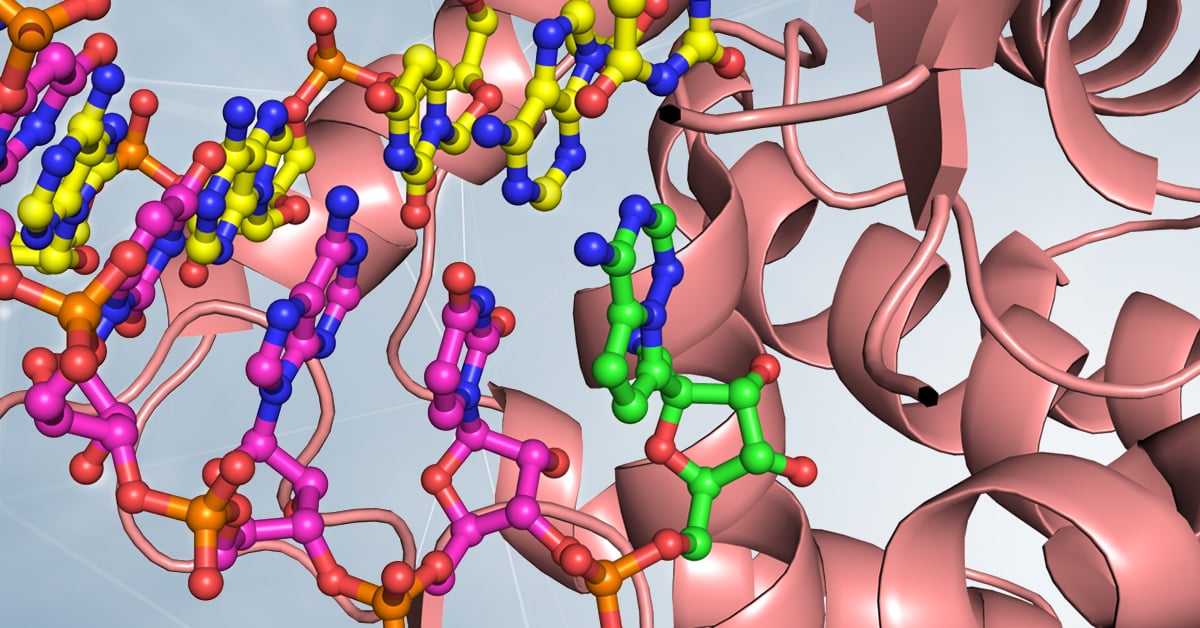
La struttura di RdRP da SARS-CoV-2, composta di tre subunità proteiche a formare un eterotrimero attivo, catturata tramite cryo-EM nel processo di replicazione del RNA, visibile nella parte sinistra. Il cuore del macchinario RdRP è evidenziato sulla destra, dove in verde è visualizzata la forma attiva del farmaco Remdesivir, incorporata nella copia del materiale genetico del virus. L’aggiunta del farmaco alla copia del RNA impedisce la copiatura completa del materiale genetico, e i dettagli fini del meccanismo sono ancora oggetto di dibattito. Gentile concessione del Dott. Degano.
Questa struttura ha dimostrato che Remdesivir agisce in un modo del tutto particolare, non tanto inceppando RdRP, ma inserendosi nella copia del RNA che viene creata e lì agendo come un segnale di “stop”, impedendo che il lavoro della RNA polimerasi possa proseguire.
Ancora più interessante, la struttura ha mostrato che il punto in cui si lega Remdesivir a RdRP prima di essere incorporato nel RNA ha delle caratteristiche ben specifiche che possono essere sfruttate dai chimici per sintetizzare una molecola antivirale ancora più efficiente.
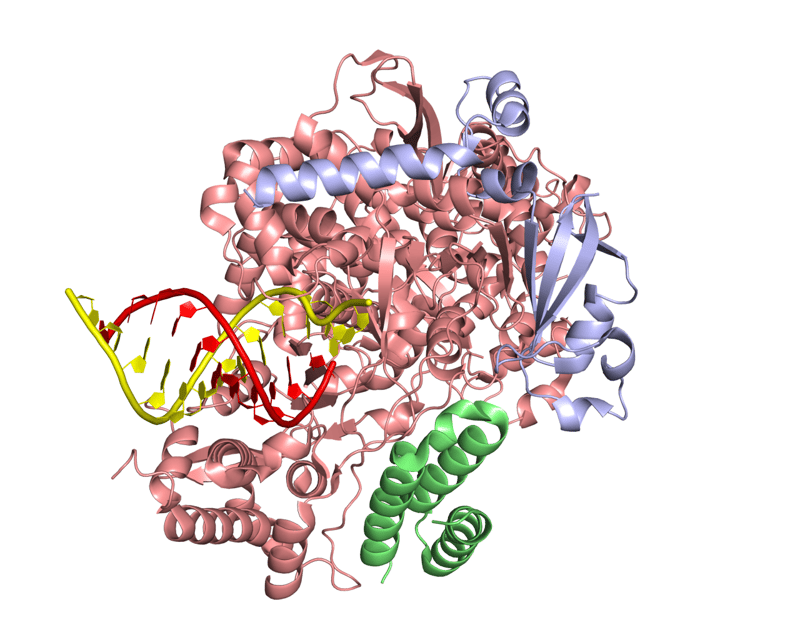
La struttura della RNA polimerasi di SARS-CoV-2, composta di tre subunità proteiche a formare un eterotrimero attivo. Gentile concessione del Dott. Degano.
Due ulteriori strutture determinate da altri gruppi in condizioni differenti hanno offerto altre informazioni sul meccanismo di azione dell’antivirale e su come ottenerne di migliori [2],[3].
Al momento, esiste già un composto chiamato EIDD-2801 che è fino a 10 volte più potente di Remdesivir nel bloccare la replicazione di SARS-CoV-2 nelle cellule in coltura [4]. La strada per un farmaco è lunga, ma grazie alla biologia strutturale si sta procedendo a grandi passi.
Citazioni